Método Científico (Incompleto)
Juntando o método científico com o conceito de causalidade
Resultados Esperados
- Entender como testes de hipótese ligam com causalidade
- Entender o método científico
Sumário
#In:
# -*- coding: utf8
from scipy import stats as ss
import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
#In:
plt.style.use('seaborn-colorblind')
plt.rcParams['figure.figsize'] = (16, 10)
plt.rcParams['axes.labelsize'] = 20
plt.rcParams['axes.titlesize'] = 20
plt.rcParams['legend.fontsize'] = 20
plt.rcParams['xtick.labelsize'] = 20
plt.rcParams['ytick.labelsize'] = 20
plt.rcParams['lines.linewidth'] = 4
#In:
plt.ion()
#In:
def despine(ax=None):
if ax is None:
ax = plt.gca()
# Hide the right and top spines
ax.spines['right'].set_visible(False)
ax.spines['top'].set_visible(False)
# Only show ticks on the left and bottom spines
ax.yaxis.set_ticks_position('left')
ax.xaxis.set_ticks_position('bottom')
Introdução
Aqui, vamos brincar um pouco com dados onde posso falar algo de causalidade. Isto é, foi feito um experimento controlado e randomizado. Note que minha ferramenta é a mesma de antes, permutação, porém a forma que os dados foram coletados mudara. Esta é a diferença.
Abaixo tenho uma função simples que permuta uma coluna de um dataframe. Vamos usar ela para implementar o nosso teste de permutação. Este exemplo é bem similar ao teste de permutação já feito. Use o mesmo para revisar!
#In:
def permuta(df, coluna):
'''
Permuta um dataframe com base e uma coluna categórica.
Este código é mais lento pois cria uma cópia.
Parâmetros
----------
df: o dataframe
coluna: uma coluna categórica
Retorna
-------
um novo df permutado
'''
novo = df.copy() # Cópia dos dados
dados = df[coluna].copy() # Copia da coluna, evitar um warning pandas. Deve ter forma melhor de fazer.
np.random.shuffle(dados) # Faz o shuffle
novo[coluna] = dados # Faz overwrite da coluna
return novo
Dados
O DataFrame consiste de dois grupos. Um de controle, outro de tratamento. No primeiro, foi medicado placebo. No segundo, foi utilizado um novo medicamento. Quando o resultado é 1, dizemos que houve melhoria nos pacientes.
#In:
df = pd.read_csv('https://media.githubusercontent.com/media/icd-ufmg/material/master/aulas/13-CausalidadeRCT/bta.csv')
df.head()
| Group | Result | |
|---|---|---|
| 0 | Control | 1.0 |
| 1 | Control | 1.0 |
| 2 | Control | 0.0 |
| 3 | Control | 0.0 |
| 4 | Control | 0.0 |
#In:
control = df.query('Group == "Control"')
control.head()
| Group | Result | |
|---|---|---|
| 0 | Control | 1.0 |
| 1 | Control | 1.0 |
| 2 | Control | 0.0 |
| 3 | Control | 0.0 |
| 4 | Control | 0.0 |
#In:
medicados = df.query('Group != "Control"')
medicados.head()
| Group | Result | |
|---|---|---|
| 16 | Treatment | 1.0 |
| 17 | Treatment | 1.0 |
| 18 | Treatment | 1.0 |
| 19 | Treatment | 1.0 |
| 20 | Treatment | 1.0 |
Ao somar o resultado de cada caso, vejo quantos melhoraram. Note que o valor é bem maior nos medicados.
#In:
control['Result'].sum()
2.0
#In:
medicados['Result'].sum()
9.0
Como os dados são 1/0, a média aqui vira uma proporção. Cada observação, $x_i \in {0, 1}$. 0 é quando não temos um efeito positivo e 1 quando temos.
\[\sum_{i=1}^{N} x_i/n\]#In:
control['Result'].mean()
0.125
#In:
medicados['Result'].mean()
0.6
Aqui tenho o efeito nos dados reais, mensurados em abs.
#In:
abs(medicados['Result'].mean() - control['Result'].mean())
0.475
#In:
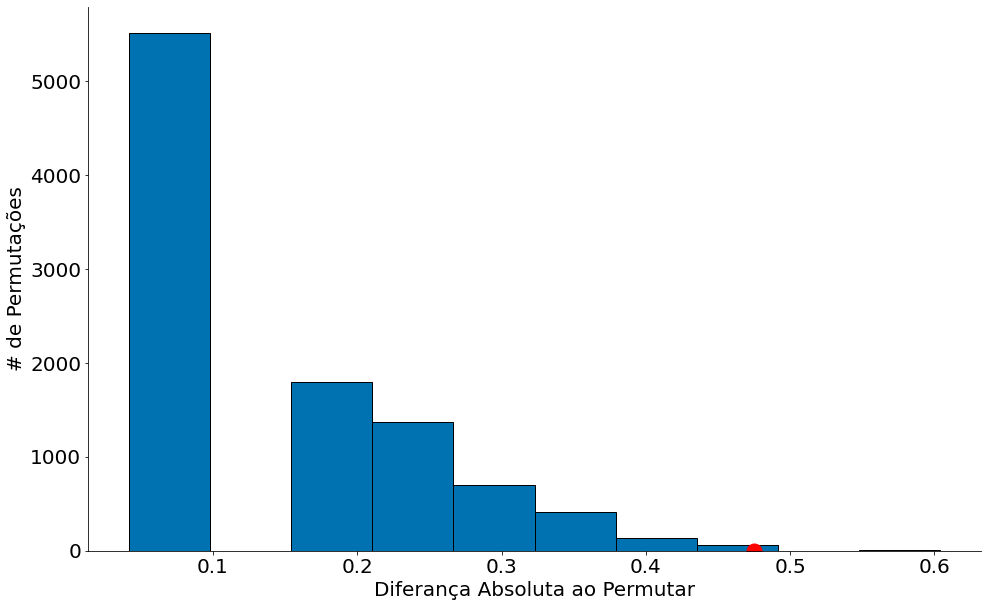
tobs = abs(medicados['Result'].mean() - control['Result'].mean())
#In:
tobs
0.475
Dados permutados
#In:
p1 = permuta(df, 'Group')
p1.head()
| Group | Result | |
|---|---|---|
| 0 | Control | 1.0 |
| 1 | Control | 1.0 |
| 2 | Treatment | 0.0 |
| 3 | Treatment | 0.0 |
| 4 | Control | 0.0 |
#In:
p1.query('Group == "Control"').mean()
Result 0.4375
dtype: float64
#In:
p1.query('Group == "Treatment"').mean()
Result 0.266667
dtype: float64
#In:
df.head()
| Group | Result | |
|---|---|---|
| 0 | Control | 1.0 |
| 1 | Control | 1.0 |
| 2 | Control | 0.0 |
| 3 | Control | 0.0 |
| 4 | Control | 0.0 |
Teste de Permutação abaixo.
#In:
valores = []
for _ in range(10000):
novo = permuta(df, 'Group')
controle = novo.query('Group == "Control"')['Result']
medicados = novo.query('Group != "Control"')['Result']
valores.append(abs(controle.mean() - medicados.mean()))
#In:
valores = np.array(valores)
#In:
valores
array([0.34583333, 0.0875 , 0.04166667, ..., 0.04166667, 0.3 ,
0.21666667])
#In:
bins = np.arange(0.15, 0.75, 0.05)
print(bins)
plt.hist(valores, bins=10, edgecolor='k')
plt.ylabel('# de Permutações')
plt.xlabel('Diferança Absoluta ao Permutar')
plt.plot([tobs], [0], 'ro', ms=15)
despine()
[0.15 0.2 0.25 0.3 0.35 0.4 0.45 0.5 0.55 0.6 0.65 0.7 ]
#In:
valor_p = (valores > tobs).mean()
valor_p
0.0011
Diferença de outros exemplos
Neste exemplo foi feito uma intervenção. Isto é, medicamos parte dos dados. Não estamos observando dados apenas. Por isto um exemplo como este é causal, vemos um efeito real em um experimento controlado! Mais importante do que isto, em 2011 foi averiguado que este pequeno estudo é um dos mais corretos quando se trata de dor crônica lombar! O ferramental aqui foram testes simples + uma boa amostra!